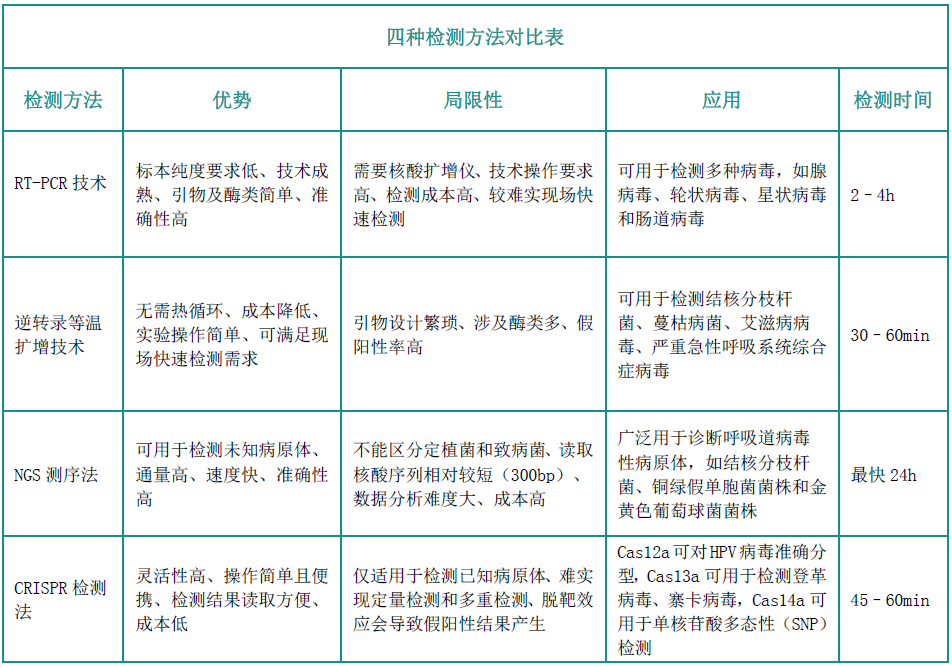
傳染病檢測有兩種方式:檢測病原體或者檢測人體為了抵抗病原體而產生的抗體。其中病原體檢測可通過抗原檢測或者核酸檢測方法進行,在COVID-19全球范圍內爆發傳播后,基于分子水平的核酸檢測技術逐漸成為病原體檢測的重要手段。目前,用于病原體檢測的主流技術有哪些?一起來看看!
熒光定量RT-PCR技術
熒光定量RT-PCR技術是將提取出的病毒RNA逆轉錄成cDNA,然后對合成的cDNA進行擴增,再利用熒光探針檢測擴增產物,通過擴增產物中的熒光信號來判斷樣本中是否含有病毒序列。熒光定量RT-PCR技術是世界各國疾控中心(CDC)采用的金標準,可以檢測患者樣本中微小病原體,是傳染病病毒傳播早期診斷的主要方法。
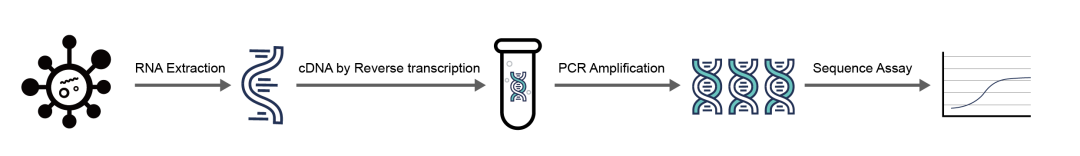
圖1 RT-PCR檢測流程圖
逆轉錄等溫擴增技術
逆轉錄等溫擴增技術是在恒定溫度下,通過添加不同活性的酶和各自特異性的引物來達到快速擴增核酸的目的。等溫擴增技術近年來發展迅速,擺脫了對熱循環儀的依賴,在現場診斷方面顯示出較強的優越性。目前主流的技術有環介導等溫擴增(Loop-mediated isothermal amplication,LAMP)、連置換擴增(strand displacement amplification,SDA)、滾環核酸擴增技術(rolling cricle amplification)、依賴核酸序列擴增技術(nucleicacid sequence-based amplification,NASBA)。LAMP具有高度特異性,識別DNA/RNA靶標特定位置可高達8個,也被稱為“八倍鏡”,它是眾多等溫擴增技術中研究和應用較為成熟的,在及時檢測POCT(Point of Care Testing)中發揮重大作用。NASBA是一種簡單的等溫信號放大法,在監測和控制動物疾病傳播方面廣泛應用。
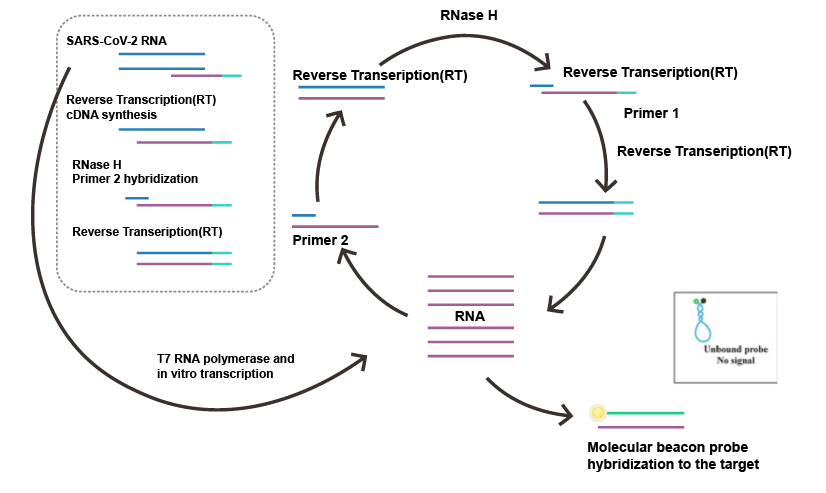
圖2 依賴核酸序列擴增技術(NASBA)檢測SARS-CoV-2病毒流程圖
NGS測序法
NGS測序法常用的原理是橋式PCR擴增,通過模板DNA分子的化學修飾,將其錨定在納米孔或者微載體芯片上,利用堿基互補配對原則,通過采集熒光標記信號或化學反應信號,實現堿基序列的解讀。NGS一次性可完成幾十萬至幾百萬條序列的測定,可在基因組水平對未知病毒進行從頭測序,獲得病原體的基因序列。與RT-PCR相比,NGS可以提供更多的病毒序列和分型信息,是識別未知病原體的有力工具。
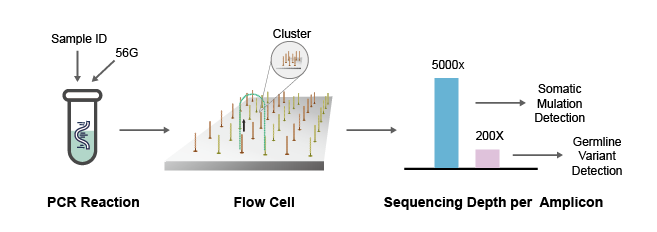
圖3 NGS測序法檢測病毒流程圖
CRISPR檢測法
CRISPR檢測法的診斷機制與基因編輯相似,在sgRNA的引導下,CRISPR-Cas蛋白識別靶標序列后,Cas12a、Cas13a和Cas14a內切酶被激活,對靶標核酸進行切割,產生熒光信號,達到檢測目的。相對于qPCR等溫擴增技術而言,CRIPSR檢測技術采取等溫擴增與Cas酶的反式切割結合的方法進行檢測,借助sgRNA的特異性識別,即提高等溫擴增后的特異性,又實現了現場快速檢測(POCT)。CRISPR診斷方法被稱為下一代分子診斷技術,有望引起體外診斷領域的新變革。
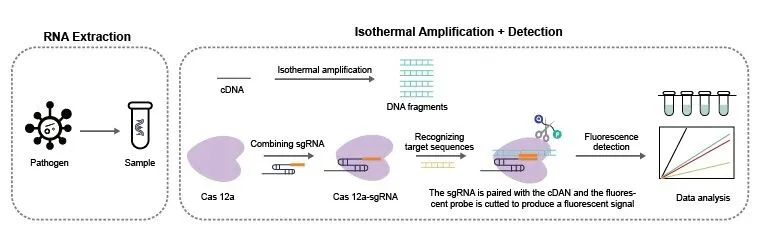
圖4 CRISPR-Cas12a系統檢測病毒流程圖
如何選擇檢測方法?
泓迅生物
引物探針設計與合成
無論選擇哪種檢測方法,都離不開核心原材料引物探針,高質量的特異性引物探針可大大提高實驗效率、檢測準確性。泓迅生物為您提供高質量引物探針設計與合成!精準的引物探針設計,先進的制造工藝和高效的交付周期,助您迅速開展病原體檢測。我們提供qPCR探針、FISH探針、分子信標探針、NGS等不同類型的核酸探針,還可為您提供定制化引物探針設計與合成服務,滿足您在分子診斷領域的各種應用需求。
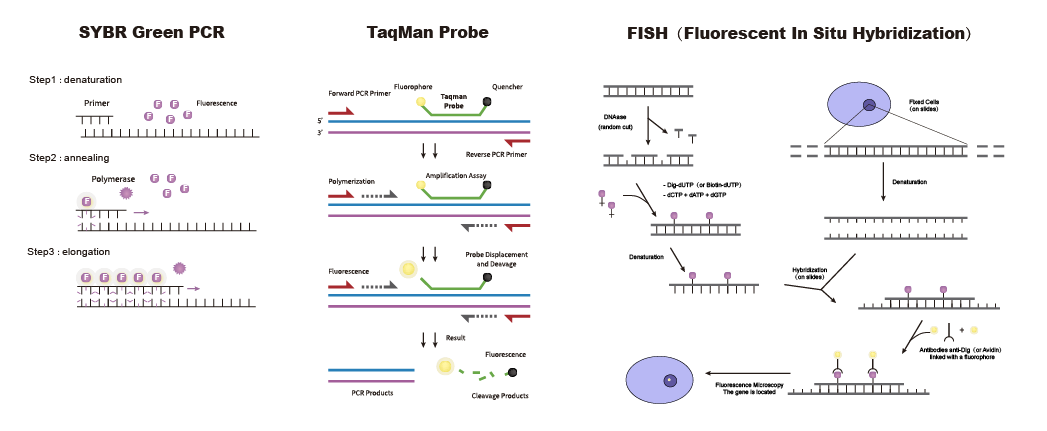
圖5三種探針示意圖
泓迅案例
引物合成是由單個堿基順次連接合成。每處堿基的合成需要完成脫保護、縮合連接、乙酰化封閉、氧化穩定四個步驟,加之化學反應自身的效率問題,最終會導致引物產物無法達到100%的純度。所以,化學合成的效率直接影響粗產物的純度,同時也限制了合成引物的長度(一般超過150nt的引物推薦使用基因合成或其它分子生物學的方法制備)。
泓迅生物升級純化工藝,優化試劑配比,最大限度的分離小分子熒光、失敗的小分子片段和目標產物,我們的引物探針產品純化效果優于市場平均水平,最高純度可達98%。
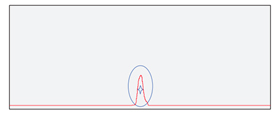
|
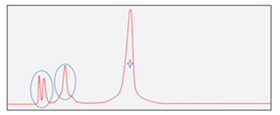
|
| 常規純化后的探針產物 | 泓迅升級純化工藝后的探針產物 |
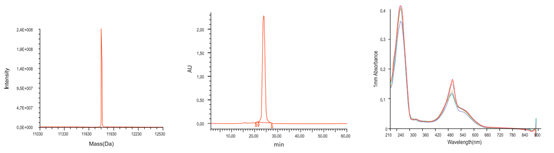
質檢結果顯示,泓迅生物制備的高質量探針,MS檢測于理論分子量相同且無N±峰,HPLC檢測純度高達98%以上,熒光全波長掃描與對應參數一致。
References
【1】Hu WS, Hughes SH. HIV-1 Reverse Transcription. Cold Spring Harb Perspect Med. 2012 Oct 1;2(10):a006882. doi: 10.1101/cshperspect.
【2】Sun Y, Yu L, Liu C, Ye S, Chen W, Li D, Huang W. One-tube SARS-CoV-2 Detection Platform Based on RT-RPA and CRISPR/Cas12a. J Transl Med. 2021 Feb 16;19(1):74. doi: 10.1186/s12967-021-02741-5.
【3】Kia V, Tafti A, Paryan M, Mohammadi-Yeganeh S. Evaluation of Real-time NASBA Assay for the Detection of SARS-CoV-2 Compared with Real-time PCR. Ir J Med Sci. 2022 Jun 6;1–7. doi: 10.1007/s11845-022-03046-2
【4】Deurenberg RH, Bathoorn E, Chlebowicz MA, etc. Application of Next Generation Sequencing in Clinical Microbiology and Infection Prevention. J Biotechnol. 2017 Feb 10;243:16-24. doi: 10.1016/j.
【5】Soroka M, Wasowicz B, Rymaszewska A. Loop-Mediated Isothermal Amplification (LAMP): The Better Sibling of PCR? Cells. 2021 Jul 29;10(8):1931. doi: 10.3390/cells10081931.
【6】 Nowrousian M. Next-generation Sequencing Techniques for Eukaryotic Microorganisms: Sequencing-based Solutions to Biological Problems. Eukaryot Cell. 2010 Sep;9(9):1300-10. doi: 10.1128/EC.00123-10.
【7】 Ortiz-Cartagena C, Fernández-García L, Blasco L, etc. Reverse Transcription-Loop-Mediated Isothermal Amplification-CRISPR-Cas13a Technology as a Promising Diagnostic Tool for SARS-CoV-2. Microbiol Spectr. 2022 Sep 28:e0239822. doi: 10.1128/spectrum.02398-22.
 Syno?C 引物合成
Syno?C 引物合成 RNA合成
RNA合成 mRNA合成
mRNA合成 Syno?GS 基因合成
Syno?GS 基因合成 載體構建
載體構建 高通量及DNA文庫構建
高通量及DNA文庫構建 CRISPR基因編輯平臺
CRISPR基因編輯平臺 病毒包裝
病毒包裝 基因測序及分析
基因測序及分析 重組蛋白表達平臺
重組蛋白表達平臺 抗體工程平臺
抗體工程平臺 多肽服務
多肽服務 生物信息學分析與設計
生物信息學分析與設計 CRISPR文庫
CRISPR文庫 ProXpress蛋白快速檢測
ProXpress蛋白快速檢測 CRISPR 質粒
CRISPR 質粒





















