“進(jìn)化是生命的標(biāo)志”——由基因突變和自然環(huán)境選擇共同編織的漫長(zhǎng)史詩(shī)。在當(dāng)今的生命科學(xué)領(lǐng)域,我們正嘗試通過(guò)定向進(jìn)化,來(lái)模仿并加速這一過(guò)程。
那么,如何加速這一進(jìn)化過(guò)程?答案就藏在基因突變文庫(kù)中。
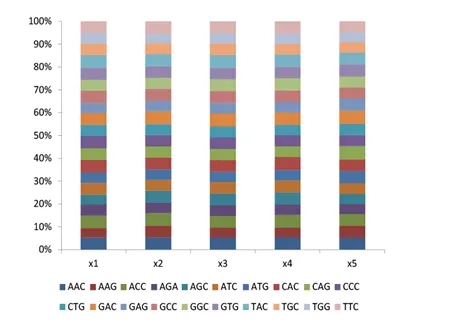
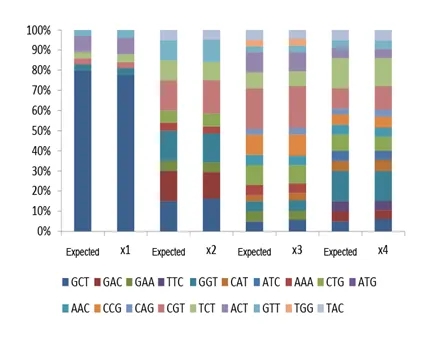
Trimer組合突變文庫(kù)以氨基酸作為一個(gè)單元進(jìn)行突變,以擴(kuò)大突變范圍。通過(guò)針對(duì)序列進(jìn)行設(shè)計(jì),可以靈活指定氨基酸的種類(lèi)和比例,避免不必要的同義密碼子和氨基酸,解決了密碼子偏移、移碼突變和控制終止密碼子摻入等問(wèn)題,同時(shí)顯著提高了文庫(kù)的均一性、覆蓋性和多樣性。
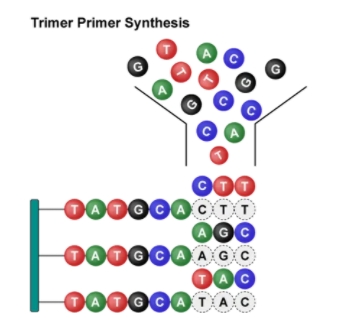
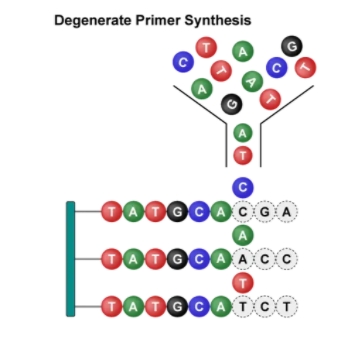
定點(diǎn)飽和突變文庫(kù)(Site-Saturation Mutagenesis Library)和Trimer組合突變文庫(kù)(Combinatorial Mutagenesis Library)是兩種各具特色的工具。前者實(shí)現(xiàn)單點(diǎn)氨基酸的精確突變,后者實(shí)現(xiàn)多點(diǎn)多種氨基酸的精確突變。它們?nèi)缤M(jìn)化的加速器,讓我們能夠更精準(zhǔn)、更高效地探索生命的奧秘。
定制化突變文庫(kù)的應(yīng)用
蛋白定向進(jìn)化
-
蛋白質(zhì)工程:通過(guò)精確調(diào)控目標(biāo)蛋白關(guān)鍵氨基酸的突變,構(gòu)建具有特定功能或性質(zhì)的蛋白質(zhì)變體,調(diào)節(jié)蛋白質(zhì)活性、穩(wěn)定性和親和性,以適應(yīng)不同應(yīng)用場(chǎng)景需求。
-
新功能發(fā)現(xiàn):引入多樣氨基酸變異以發(fā)現(xiàn)新蛋白功能,如催化新反應(yīng)、結(jié)合新配體等,促進(jìn)蛋白功能性篩選和進(jìn)化。
-
抗藥性研究:模擬藥物壓力可篩選對(duì)抗藥物具耐受性的突變體,揭示藥物抗性機(jī)制,為優(yōu)化藥物治療提供指導(dǎo)。
抗體開(kāi)發(fā)
-
增強(qiáng)親和力:引入特定氨基酸變異可改變抗體與靶標(biāo)分子結(jié)合親和力,提高治療效果和選擇性。
-
改善穩(wěn)定性:調(diào)整抗體結(jié)構(gòu)可增強(qiáng)體內(nèi)穩(wěn)定性和半衰期,延長(zhǎng)藥效持續(xù)時(shí)間、減少劑量頻率,提高患者治療便利性。
-
降低免疫原性:避免或減少抗體與人類(lèi)免疫系統(tǒng)相抗原的位點(diǎn),降低免疫原性和產(chǎn)生不良反應(yīng)風(fēng)險(xiǎn),使抗體更適合臨床治療。
藥物靶點(diǎn)篩選
-
靶點(diǎn)發(fā)現(xiàn):精準(zhǔn)控制和定向突變已知靶點(diǎn)特定區(qū)域,產(chǎn)生具有新結(jié)構(gòu)和功能特性的變體,為新藥物靶點(diǎn)發(fā)現(xiàn)提供工具和平臺(tái)。
-
靶點(diǎn)驗(yàn)證:整突變體中各氨基酸比例和組合,覆蓋和測(cè)試多種功能性質(zhì),全面評(píng)估不同突變體的活性、親和性、穩(wěn)定性等重要性質(zhì)。
-
藥物篩選:高通量篩選大量不同序列蛋白質(zhì)變體,找到與特定藥物高親和力的靶點(diǎn),加速藥物篩選,提高藥物發(fā)現(xiàn)效率。
泓迅生物領(lǐng)先設(shè)計(jì)和先進(jìn)制造提供Trimer文庫(kù)一站式解決方案,已成功交付了數(shù)百個(gè)高品質(zhì)、定制化的突變文庫(kù)。精準(zhǔn)控制CDR區(qū)域的每個(gè)突變位點(diǎn),調(diào)整氨基酸種類(lèi)和比例,提升文庫(kù)的準(zhǔn)確度和實(shí)驗(yàn)的成功率,正確率最高可達(dá)90%以上。降低后期篩選難度,節(jié)省時(shí)間和精力,享受高性?xún)r(jià)比服務(wù)。

References
[1] Prassler, Josef, et al. "HuCAL PLATINUM, a synthetic Fab library optimized for sequence diversity and superior performance in mammalian expression systems." Journal of molecular biology 413.1 (2011): 261-278.
[2] Chembath, Anupama, et al. "Nondegenerate Saturation Mutagenesis: Library Construction and Analysis via MAX and ProxiMAX Randomization." Directed Evolution: Methods and Protocols. New York, NY: Springer US, 2022. 19-41.
 Syno?C 引物合成
Syno?C 引物合成 RNA合成
RNA合成 mRNA合成
mRNA合成 Syno?GS 基因合成
Syno?GS 基因合成 載體構(gòu)建
載體構(gòu)建 高通量及DNA文庫(kù)構(gòu)建
高通量及DNA文庫(kù)構(gòu)建 CRISPR基因編輯平臺(tái)
CRISPR基因編輯平臺(tái) 病毒包裝
病毒包裝 基因測(cè)序及分析
基因測(cè)序及分析 重組蛋白表達(dá)平臺(tái)
重組蛋白表達(dá)平臺(tái) 抗體工程平臺(tái)
抗體工程平臺(tái) 多肽服務(wù)
多肽服務(wù) 生物信息學(xué)分析與設(shè)計(jì)
生物信息學(xué)分析與設(shè)計(jì) CRISPR文庫(kù)
CRISPR文庫(kù) ProXpress蛋白快速檢測(cè)
ProXpress蛋白快速檢測(cè) CRISPR 質(zhì)粒
CRISPR 質(zhì)粒